01 (ES). Introducción a DP0.2 (principiante)¶
Faceta del RSP: Notebook (línea-de-comandos en la terminal)
Autoría: Gloria Fonseca Alvarez
Última verificación de ejecución: 11 sep 2023
Versión de las pipelines científicas de LSST: Weekly 2023_35
Nivel de aprendizaje: principiante
Tamaño del contenedor (Container size): medium
Introducción: este tutorial es una introducción a las funcionalidades de la terminal y línea de comandos de la Plataforma Científica de Rubin (Rubin Science Plataform - RSP). Es paralelo al tutorial de Jupyter Notebook “Introducción a DP02” y muestra cómo utilizar el servicio TAP para consultar y obtener datos del catálogo; usar matplotlib para graficar los datos de un catálogo; el paquete Butler de LSST para consultar y obtener datos de imágenes; y el paquete de imágenes afwDisplay de LSST.
Este tutorial usa el conjunto de datos Vista Previa de Datos 0.2 (DP0.2 - Data Preview 0.2). Este conjunto de datos utiliza un subconjunto de las imágenes simuladas del Desafío de Datos 2 de DESC (DC2 - DESC’s Data Challenge 2), las cuales han sido reprocesadas por el Observatorio Rubin usando la versión 23 de las Pipelines Científicas del LSST (LSST Science Pipelines). Se puede encontrar más información sobre los datos simulados en el artículo científico de DC2 de DESC y en la documentación de la Divulgación de Datos de DP0.2 (DP0.2 data release).
Paso 1. Acceso a la terminal y configuración¶
1.1. Iniciar sesión en la Faceta Notebook. La terminal es un subcomponente de la Faceta Notebook.
1.2. En la ventana de lanzadores debajo de “Other”, seleccionar la “Terminal”.
1.3. Configurar el entorno del Observatorio Rubin.
setup lsst_distrib
1.4. Iniciar una sesión interactiva de Python.
python
Paso 2. Importar paquetes¶
Este tutorial hace uso de numerosos paquetes que serán utilizados con frecuencia cuando se interactúe con los datos de catálogos y de las imágenes.
2.1. Importar paquetes generales de python.
import numpy
import matplotlib
import matplotlib.pyplot as plt
2.2. Importar paquetes necesarios para acceder a los datos de los catálogos.
import pandas
pandas.set_option('display.max_rows', 1000)
from lsst.rsp import get_tap_service, retrieve_query
2.3. Importar paquetes necesarios para acceder a imágenes.
import lsst.daf.butler as dafButler
import lsst.geom
import lsst.afw.display as afwDisplay
Paso 3. Obtener datos usando TAP para 10 objetos¶
El protocolo de Acceso a Datos Tabulados TAP (Table Access Protocol) provee acceso estandarizado a los datos de los catálogos para exploración, búsqueda y acceso. La documentación completa de TAP es provista por la Alianza Internacional del Observatorio Virtual (IVOA - International Virtual Observatory Alliance). El servicio TAP utiliza un lenguaje de consultas similar a SQL (Structured Query Language - lenguaje de consulta estructurado) denominado ADQL (Astronomical Data Query Language - lenguaje de consulta de datos astronómicos). La documentación de ADQL incluye más información sobre sintaxis y palabras clave.
Aviso: no todas las funcionalidades de ADQL están soportadas por RSP para la Vista Previa de Datos 0 (DP0).
Este ejemplo utiliza el Catálogo de Objetos DP0.2, que contiene fuentes detectadas en las imágenes coagregadas (también llamadas imágenes apiladas, combinadas o deepCoadd).
3.1. Iniciar el servicio TAP.
service = get_tap_service("tap")
3.2. Definir las coordenadas para una búsqueda en un cono centrado alrededor de la región cubierta por la simulación DC2 (RA,Dec = 62,-37).
use_center_coords = "62, -37"
3.3. Crear una consulta llamada my_adql_query para obtener las coordenadas y las magnitudes g, r, i para objetos contenidos en un radio de 0.5 grados de las coordenadas centrales.
my_adql_query = "SELECT TOP 10 coord_ra, coord_dec, detect_isPrimary, " + \
"r_calibFlux, r_cModelFlux, r_extendedness " + \
"FROM dp02_dc2_catalogs.Object " + \
"WHERE CONTAINS(POINT('ICRS', coord_ra, coord_dec), " + \
"CIRCLE('ICRS', " + use_center_coords + ", 0.5)) = 1 "
3.4. Obtener y visualizar los resultados de la consulta para 10 objetos.
results = service.search(my_adql_query)
results_table = results.to_table()
print(results_table)
3.5. Convertir flujos en magnitudes.
Los catálogos de objetos y fuentes almacenan sólo flujos. Hay cientos de columnas relacionadas a flujos, y almacenarlas también como magnitudes sería redundante, y un desperdicio de espacio. Todas las unidades de flujos son nanojanskys (nJy). Para convertir nJy a magnitudes AB usar: mAB = -2.5log(fnJy) + 31.4.
Agregar columnas de magnitudes después de obtener columnas de flujo.
results_table['r_calibMag'] = -2.50 * numpy.log10(results_table['r_calibFlux']) + 31.4
results_table['r_cModelMag'] = -2.50 * numpy.log10(results_table['r_cModelFlux']) + 31.4
Visualizar la tabla de resultados incluyendo las magnitudes.
print(results_table)
Paso 4. Obtener datos usando TAP para 10,000 objetos¶
Para obtener las columnas correspondientes a flujos como magnitudes con una consulta ADQL, se puede hacer lo siguiente: scisql_nanojanskyToAbMag(g_calibFlux) as g_calibMag, y las columnas de errores de magnitudes se pueden obtener con: scisql_nanojanskyToAbMagSigma(g_calibFlux, g_calibFluxErr) as g_calibMagErr.
4.1. Obtener las magnitudes de las bandas g, r e i para 10000 objetos puntuales.
A la búsqueda en un cono realizada en la consulta previa, agregarle como restricciones que detect_isPrimary sea True (esto excluirá fuentes “hijas” producto de la separación - deblending), que el flujo calibrado sea mayor que 360 nJy (aproximadamente magnitud 25), y que los parámetros de extensión sean 0 (fuentes puntuales).
results = service.search("SELECT TOP 10000 coord_ra, coord_dec, "
"scisql_nanojanskyToAbMag(g_calibFlux) as g_calibMag, "
"scisql_nanojanskyToAbMag(r_calibFlux) as r_calibMag, "
"scisql_nanojanskyToAbMag(i_calibFlux) as i_calibMag, "
"scisql_nanojanskyToAbMagSigma(g_calibFlux, g_calibFluxErr) as g_calibMagErr "
"FROM dp02_dc2_catalogs.Object "
"WHERE CONTAINS(POINT('ICRS', coord_ra, coord_dec), "
"CIRCLE('ICRS', "+use_center_coords+", 1.0)) = 1 "
"AND detect_isPrimary = 1 "
"AND g_calibFlux > 360 "
"AND r_calibFlux > 360 "
"AND i_calibFlux > 360 "
"AND g_extendedness = 0 "
"AND r_extendedness = 0 "
"AND i_extendedness = 0")
4.2. Almacenar los datos como un objeto dataframe (marco de datos) de Pandas.
results_table = results.to_table()
data = results_table.to_pandas()
Paso 5. Hacer un diagrama color-magnitud¶
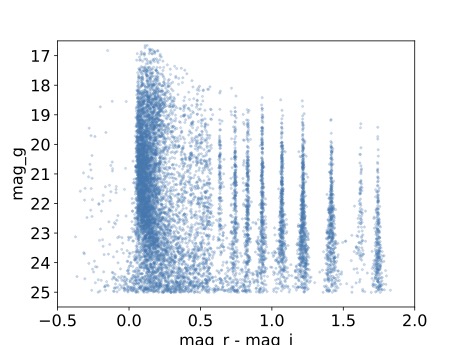
5.1. Graficar el color (magnitudes r-i) vs magnitud g.
plt.plot(data['r_calibMag'].values - data['i_calibMag'].values,
data['g_calibMag'].values, 'o', ms=2, alpha=0.2)
5.2. Definir las etiquetas y los límites de los ejes.
plt.xlabel('mag_r - mag_i', fontsize=16)
plt.ylabel('mag_g', fontsize=16)
plt.xticks(fontsize=16)
plt.yticks(fontsize=16)
plt.xlim([-0.5, 2.0])
plt.ylim([25.5, 16.5])
5.3. Guardar el gráfico en formato pdf.
plt.savefig('color-magnitude.pdf')
Utilizar el navegador de archivos en el lado izquierdo de la Faceta Notebook para navegar hasta el archivo “color-magnitude.pdf”. Hacer doble clic sobre el nombre de archivo para abrirlo y ver el gráfico.
Paso 6. Obtener los datos de imágenes usando Butler¶
Los dos tipos de imágenes más comunes con los que van a interactuar quienes tengan acceso a DP0 son calexps y deepCoadds.
calexp: una única imagen en un único filtro.
deepCoadd: una combinación de imágenes individuales apiladas en profundidad o coagregadas.
Las Pipelines Científicas LSST (Science Pipelines) procesan y almacenan imágenes en regiones y parcelas. Para obtener y mostrar una imagen en una coordenada deseada, se debe especificar el tipo de imagen, región (tract) y parcela (patch).
región (tract): una porción del cielo dentro de la teselación del cielo completo (mapa del cielo) de LSST (LSST all-sky tessellation); dividido en parcelas.
parcela (patch): una subregión cuadrilátera de una región, de un tamaño que puede almacenarse fácilmente en la memoria de una computadora de escritorio.
Butler - que en inglés significa mayordomo - (documentación de butler) es un paquete de software de las Pipelines Científicas de LSST para obtener datos de LSST sin necesidad de conocer su ubicación o formato. Además, Butler también puede ser utilizado para explorar y descubrir qué datos existen. Otros tutoriales muestran la funcionalidad completa de Butler.
6.1. Definir una configuración y colección de Butler.
butler = dafButler.Butler('dp02', collections='2.2i/runs/DP0.2')
6.2. Definir las coordenadas de un cúmulo de galaxias conocido en DC2.
my_ra_deg = 55.745834
my_dec_deg = -32.269167
6.3. Usar lsst.geom para definir un SpherePoint (punto en la esfera) para las coordenadas del cúmulo (documentación de lsst.geom).
my_spherePoint = lsst.geom.SpherePoint(my_ra_deg*lsst.geom.degrees, my_dec_deg*lsst.geom.degrees)
print(my_spherePoint)
6.4. Obtener el mapa del cielo de DC2 (skymap) (documentación de skymap) e identificar la región y parcela (tract y patch).
skymap = butler.get('skyMap')
tract = skymap.findTract(my_spherePoint)
patch = tract.findPatch(my_spherePoint)
my_tract = tract.tract_id
my_patch = patch.getSequentialIndex()
print('my_tract: ', my_tract)
print('my_patch: ', my_patch)
6.5. Utilizar Butler para obtener la imagen deepCoadd en la banda i.
dataId = {'band': 'i', 'tract': my_tract, 'patch': my_patch}
my_deepCoadd = butler.get('deepCoadd', dataId=dataId)
Paso 7. Visualizar la imagen¶
Los datos de imágenes recuperados con Butler se pueden visualizar de muchas formas distintas.
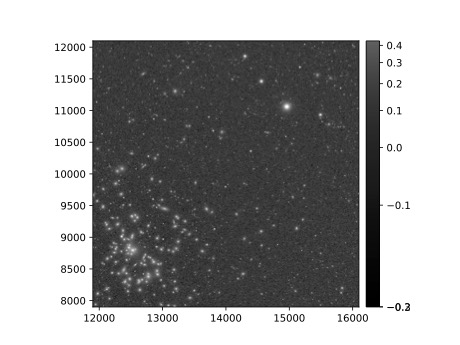
7.1. Visualizar la imagen usando afwDisplay (documentación de afwDisplay).
afwDisplay.setDefaultBackend('matplotlib')
fig = plt.figure(figsize=(10, 8))
afw_display = afwDisplay.Display(1)
afw_display.scale('asinh', 'zscale')
afw_display.mtv(my_deepCoadd.image)
plt.gca().axis('on')
plt.savefig('my_deepCoadd.pdf')
Utilizar el navegador de archivos en el lado izquierdo de la Faceta Notebook para navegar hasta el archivo “my_deepCoadd.pdf” Hacer doble clic sobre el nombre de archivo para abrirlo y ver el gráfico.
7.2. Visualizar la imagen usando Firefly (documentación de Firefly).
afwDisplay.setDefaultBackend('firefly')
afw_display = afwDisplay.Display(frame=1)
afw_display.mtv(my_deepCoadd)
Opcional: para una demostración de la interfaz interactiva de Firefly, revisar “03b Visualización de imágenes con Firefly” del Notebook tutorials.
7.3. Al terminar, salir de python para regresar a la línea de comando normal.
exit()